生物学和医学研究人员使用空间转录组学 (ST) 技术来检测细胞中的转录水平、预测细胞类型并构建组织的三维 (3D) 结构。然而,当有多个组织切片需要使用最先进的工具包进行联合分析时,这种分析可能会很困难。对于研究人员来说,手动组装切片并构建 3D 结构是一项挑战。
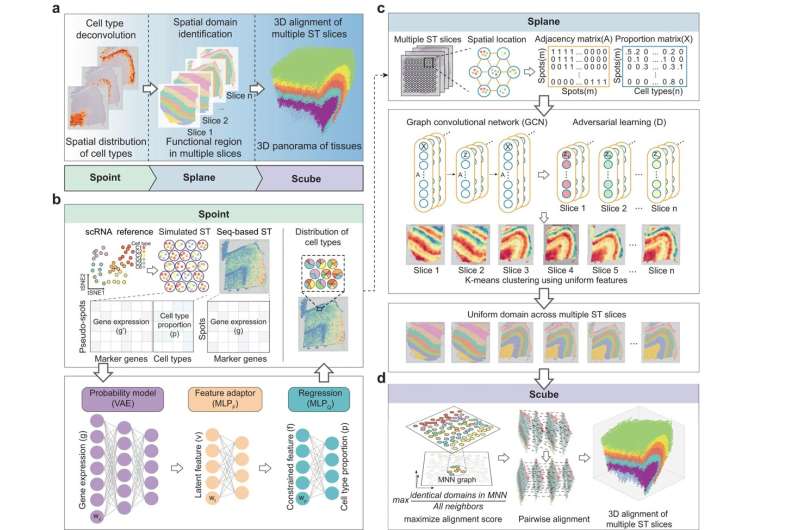
为了解决这个问题,中国科学院科技大学曲昆教授领导的研究小组开发了一种新的空间架构表征方法,即(SPACEL)通过Spoint、Splane、Scube三个模块,SPACEL可以自动构建组织的三维全景图。
他们的研究结果是发布在自然通讯。
这三个模块针对 ST 分析中的三个主要任务而设计。Sprint 可以执行细胞类型反卷积,以预测. 模拟伪点、神经网络建模和表达谱统计恢复相结合,保证了预测的稳健性和准确性。
Splane 采用图卷积网络方法和对抗学习算法,通过联合分析多个 ST 切片来识别特殊域,而 Scube 则自动对齐切片并构建堆叠的 3D 架构通过三个模块,从原始数据构建组织的3D架构。
研究人员将SPACEL应用于11个ST数据集,共计156张切片,过程中涉及10X Visium、STARmap、MERFISH、Stereo-seq、Spatial Transcriptomics等技术;在预测细胞类型分布、识别空间区域和重建三维组织结构三个核心分析任务中,SPACEL在细胞类型反卷积方面展现出了优于其他算法的性能。
这项研究为ST数据处理和分析提供了宝贵的集成工具包,有利于进一步利用ST技术进行研究。
中国科学技术大学提供
引用: 一种基于深度学习的空间转录组学分析工具包(2024 年 1 月 2 日)于 2024 年 5 月 24 日检索自 https://webbedxp.com/zh-CN/science/jamaal/news/2024-01-deep-learning-based-analysis-toolkit-spatial-transcriptomics.html
本文件受版权保护。除出于私人学习或研究目的的合理使用外,未经书面许可不得复制任何部分。内容仅供参考。









