合成新蛋白质——生物生命——是一个潜力巨大的科学领域,新开发的人工智能模型有望为新蛋白质创建指令,远远超出自然界中发现的蛋白质。
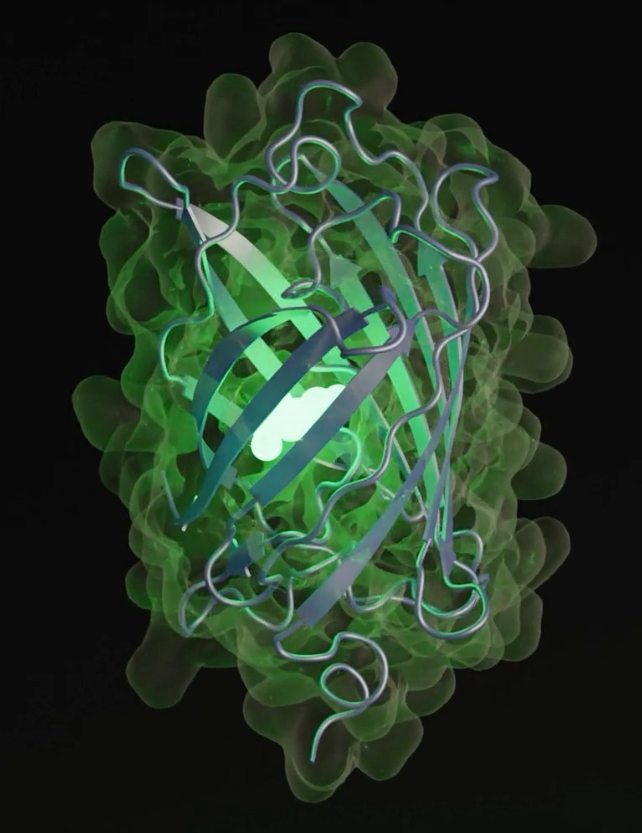
美国科学家使用 EvolutionaryScale Model 3 (ESM3) 合成了一种名为 esmGFP(绿色荧光蛋白)的新蛋白质,该蛋白质与其最接近的天然亲戚仅共享 58% 的材料标签RFP。
研究团队估计,这相当于人工智能处理了 5 亿年的进化,并且它为创建可设计用于特定用途的定制蛋白质或从现有蛋白质中解锁更多功能开辟了道路。
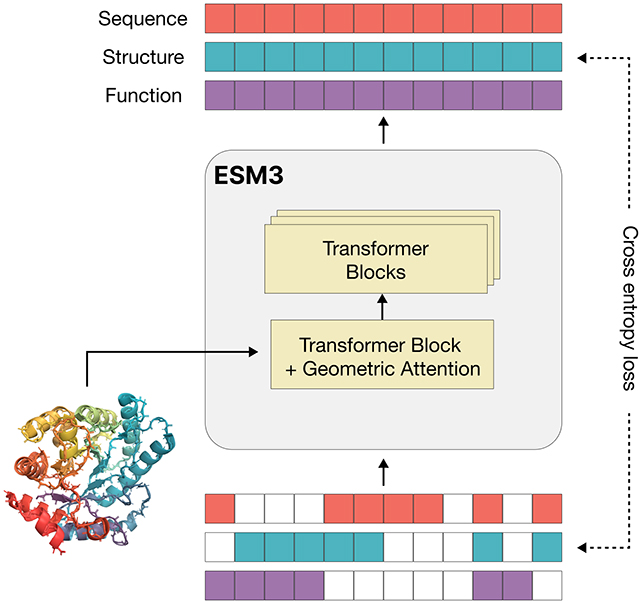
“超过三十亿年的进化已经产生了编码到天然蛋白质空间中的生物图像,”写由纽约 EvolutionaryScale 创始人 Thomas Hayes 领导的研究人员在他们发表的论文中表示。
“在这里,我们表明,根据进化数据进行大规模训练的语言模型可以生成远离已知蛋白质的功能蛋白质。”
我很高兴能分享我们一直在努力的事情@EvoscaleAI。 ESM3 是一种用于编程生物学的多模式生成屏蔽语言模型。以下是关于 ESM3 背后架构的简短讨论。 🧵https://t.co/jldHYRAPNy
— 托马斯·海耶斯 (@THayes427)2024 年 6 月 25 日
ESM3 接受了令人印象深刻的 31.5 亿个蛋白质序列(顺序为氨基酸蛋白质)、2.36 亿个蛋白质结构(它们的 3D 形状)和 5.39 亿个蛋白质注释(描述性标签)。
通过发现海量数据中的模式,人工智能模型可以了解蛋白质构建和功能中哪些有效、哪些无效,就像 ChatGPT 在阅读了数百万首人类写的诗歌后可以创作一首押韵的新诗一样。
esmGFP 的特别之处在于它的工作原理:它像其相关 tagRFP 一样具有荧光。荧光蛋白给一些海洋生物发光, 和有在。
“我们选择荧光的功能是因为它很难实现,但易于测量,而且是自然界最美丽的机制之一,”该团队写。
人工智能消除了很多尝试和错误,同时增加了探索远离我们目前所知的蛋白质的能力。
“蛋白质可以被视为存在于一个有组织的空间内,其中每种蛋白质都与其他蛋白质相邻,相距一个突变事件,”写研究人员。 “进化的结构在这个空间中表现为一个网络,通过进化在它们之间采取的路径连接所有蛋白质。”
研究小组表示,为了实现进化,每种蛋白质都必须转变为下一种蛋白质,而它所属的系统又不能失去其整体功能。语言模型可以识别这个空间中的蛋白质。
ESM3设计的蛋白质仍需要验证、合成和测试,这需要时间,但团队有信心在此取得进一步进展。在不远的将来,只需一些聪明的人工智能提示,我们就可以生产从药物到生物材料等各种用途的蛋白质。
研究人员表示:“蛋白质语言模型并不明确地在进化的物理限制内工作,而是隐式地构建了进化可能遵循的多种潜在路径的模型。”解释。
该研究发表于科学。